Pythonのパッケージ「dicom2nifti」を使ってDICOMをNIfTIに変換する
NIfTIとは
医用画像のファイルフォーマットは基本的にDICOM形式ですが、DTIやfMRIなどのいわゆる脳のアドバンスドな解析に用いられるデータはNIfTI (Neuroimaging Informatics Technology Initiative) 形式に変換して解析することが一般的なようです。「.nii」や「.nii.gz」といった拡張子がNIfTI形式に相当します。「.nii.gz」は「.nii」をgzip圧縮したフォーマットです。
DICOMはスライスごとにファイルが作成されますが、NIfTIはひとつのシーケンスごとのすべてのスライスの画像をひとつのファイルとして保存することができます。この点がNIfTIの最大のメリットかと思います。ちなみにDICOMのヘッダーはNIfTIにおいてもヘッダーとして一部保持されます。
DICOMをNIfTIに変換するためには専用のソフトウェアが必要です。調べてみたところ、先人たちがさまざまなソフトウェアを紹介してくれています。
2021年8月現時点でこれらをまとめてみますと、DICOMからNIfTIに変換するソフトウェアとして以下の3つがメジャーなようです。どのソフトウェアもGUIで操作できるので簡単に使用できます。SPMはMATLABが必要なので有料ですが、それ以外はフリーのソフトウェアです。
このように多くの便利なソフトウェアがありますが、再現可能性を追求している私はPythonだけでファイル形式変換から解析までを一貫して行いたいと考えました。Pythonのパッケージがあれば実現できそうだと思い探してみたところ、dicom2niftiというパッケージを見つけました。
githubのコミット状況を見てみると現在も開発は継続しているようにみえます。今回の記事ではdicom2niftiを用いたファイルフォーマットの変換を試みたいと思います。
方法
動作環境と使用したデータ
インストール方法
anacondaでは以下のコマンドでインストールできます。
conda install -c conda-forge dicom2nifti
実行方法
Pythonスクリプトから実行
githubのREADMEに実行方法が紹介されています。以下の2行で変換できます。
import dicom2nifti dicom2nifti.convert_directory(dicom_directory, output_folder, compression=True, reorient=True)
dicom2nift.convert_directoryの引数は
- dicom_directory
- 変換するDICOMファイルが含まれるdirectory
- output_folder
- 出力先のdirectory
- compression (True or False)
- gzip圧縮をするかどうか
- reorient (True or False)
- 画像の向きをLASに変換するかどうか
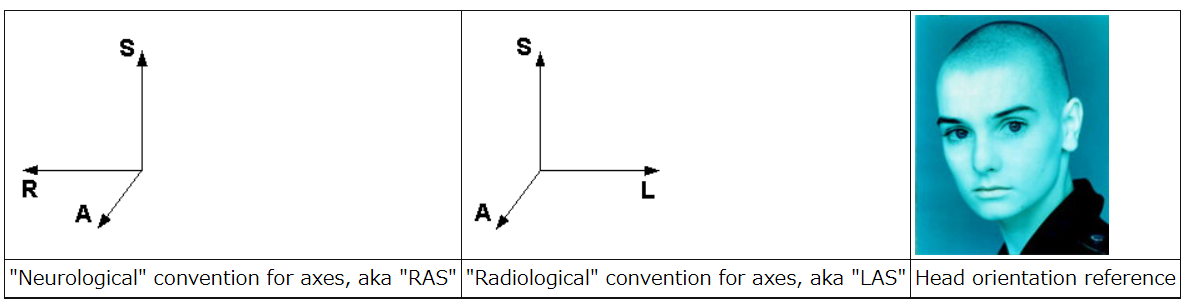
になります。僕ら「放射線科系」の人たちは画像の右側が対象の左側に対応している(LASと呼ぶらしい)のが通常の医用画像だと考えていますが、「神経科学系」の人たちは「放射線科系」の人が見ている画像の左右を逆にして表示すること(RASと呼ぶらしい)が通例なようです。画像の表示方法にも文化の違いがあるみたいですね。以下の画像がわかりやすいです。
コマンドラインから実行
コマンドラインからは以下のコマンドで実行できるようです。
dicom2nifti [-h] [-G] [-r] [-o RESAMPLE_ORDER] [-p RESAMPLE_PADDING] [-M] [-C] [-R] input_directory output_directory
各引数は
- -h (--help)
- ヘルプメッセージを表示
- -G (--allow-gantry-tilting)
- (CTの)ガントリーの傾けたデータの変換を可能にする
- -I (--allow-inconsistent-slice-increment)
- 一貫性のないスライスインクリメントデータの変換を可能にする
- -S (--allow-single-slice)
- シングルスライス(2D画像)の変換を可能にする
- -r (--resample)
- ガントリーが傾いたデータを直交画像に再サンプルしたり、スライス増分が一致しないデータを均一な画像に再サンプルする。
- -o RESAMPLE_ORDER (--resample-order RESAMPLE_ORDER)\
- リサンプリング時に使用するスプライン補間の次数 (0 -> 5) [0 = NN, 1 = LIN, ....]
- -p RESAMPLE_PADDING (--resample-padding RESAMPLE_PADDING)
- リサンプリング時に使用されるパディング値を塗りつぶし値として使用する
- -M (--allow-multiframe-implicit)
- implicit vr転送構文によるマルチフレームデータの変換を可能にする(動作保証はしていません)
- -C (--no-compression)
- gzip圧縮を無効にして、.nii.gzではなく.niiファイルを書き込む。
- -R (--no-reorientation)
- 画像のreorientationを無効にする(デフォルト:画像はLASの方向に再配置される)
のようです。こちらの詳細は詳しく書きませんが、githubを確認すれば書いていると思います。
windows環境でコマンドラインから実行する際は以下のissueが参考になりました。
簡単に説明すると、anaconda powershell promptを開いて仮想環境のdirectoryに移動して以下のコマンドを入力します。
mv .\Scripts\dicom2nifti .\Scripts\dicom2nifti_tool
その後、コマンドを実行する際は以下の通り入力します。
.\python.exe .\Scripts\dicom2nifti_tool --help # or other parameters`
結果
pythonスクリプトおよびコマンドラインからの実行はどちらもエラーがひとつでましたが、うまく変換できました。
エラーはdicom2nifti.exceptions.ConversionValidationError: TOO_FEW_SLICES/LOCALIZERと表示され、「シリーズ枚数が少ない画像はNIfTIに変換できませんでした」というエラーのようです。今回使用したデータのなかにシリーズが3枚のlocalizerがあり、それが引っ掛かったようです。ソースコードを見てみるとシリーズが3枚以下のシーケンスのデータはNIfTIに変換されない仕様になっているみたいです。少し探しましたが、これを回避する方法は見当たりませんでした。基本的に解析に使用する頭部の画像が4枚以下になることはないので、大きな問題はないと思います。
2021.8.29追記
dicom2nifti.disable_validate_slicecount()を実行しておくことで、3枚以下のシーケンスのデータもNIfTIに変換できるようになるようです。追記終わり。
2021.12.15追記
上のコードではダメでした。
import dicom2nifti.settings as settings settings.disable_validate_slicecount()
こちらのコードが正解です。追記終わり。
引数reorientをTrueにしたところ元の画像の上下が逆になりました。今回検証できたのはもともとがLASの画像に対してreorientをTrueにしても上下が逆になるだけで、Falseにすると元の向きのままだったという点のみです。元のDICOMがRASの場合であればLASに変わるのかもしれませんが、そのような画像が手元にないので検証できませんでした。
ひとこと
dicom2niftiはざっと調べたところDICOMからNIfTIへ変換できる唯一のPythonパッケージのようです。
(追記終わり)
pythonスクリプトは2行で変換できるので簡単でいいですね。
しかし、dcm2niixもしくはMRIconvertを使ったほうが自由度が高そうです。これらはNIfTIで出力する際のファイル名やdirectory構造を操作できるので、変換後のデータの整理の手間が省けそうです。なおMRIconvertはwindowsでは32bit版しかないので多くのファイルの処理をする際にはもしかしたらdcm2niixのほうが動作が早いかもしれません(これは想像です)。
まとめ
DICOMからNIfTIに変換するソフトにはdcm2niixもしくはMRIconvertを使うのがベターかもしれません。Pythonだけでどうにかしたいのであればdicom2niftiを使うのもいいかもしれません。
Motion Artifact Generator (MAGE) をPythonで実装した
楽しみにしていた以下の論文を読みました。
この論文はMAGE (motion artifact generator)と呼ばれるMRI画像のモーションアーチファクトジェネレータを開発した論文です。アーチファクトを擬似的に作成できれば、元の画像を教師画像とした深層学習モデルを作成することができます。MAGEはモデル作成時に必要なモーションアーチファクト画像を大量に作成するためのシステムだそうです。
MAGEは以下の図のようにしてモーションアーチファクト画像を作成します。MRI画像を複数のパターンでシフトさせ、それらの画像をフーリエ変換して得られた擬似k-spaceを組み合わせることでモーションアーチファクトを生成しているようです*1。詳細は論文をご覧ください。

面白そうだと思ったので、MAGEをpythonで実装してみました。今回のブログではそれを報告したいと思います。コードはgithubにあげています。
目次
動作環境
動作の検証
使用画像
今回使用した画像は、エムアールアイシミュレーションズ様が開発したBlochSolverで以前シミュレーションした人体頭部のSEのT2WI画像です。
まず、シミュレーションで得たデータを読み込みます。
import numpy as np import matplotlib.pyplot as plt from PIL import Image # 作成したmageモジュールをインポート import mage # BlochSolverでシミュレートして作成したSE画像を読み込むための関数 def sim2img(path, matrix): with open(path, 'rb') as f: d = f.read() row_data = Image.frombytes('F', (matrix[0]*2, matrix[1]), d) row_data = np.array(row_data) row_data.shape img = np.zeros(matrix, dtype=np.complex64) for col in range(matrix[1]): for row in range(matrix[0]): img[row, col] = row_data[row, col*2] + (row_data[row, (col*2+1)]*1j) return img # BlochSolverで作成したMRI画像を読み込む path = '2D_SpinEcho-3-complex.flt' kspc = sim2img(path, [256,256]) # k-spaceを表示 mage.plot_kspace(kspc)

# 強度画像を表示 t2wi = np.abs(np.fft.ifftshift(np.fft.ifft2(kspc))) plt.imshow(t2wi, cmap='gray')

MRI画像をシフトさせる
次にMRI画像をシフトさせてみます。今回は論文と同様に前後方向に-10〜10 pixel移動した画像を作成します。
# Image形式に変換 data = Image.fromarray(t2wi) # SEでモーションアーチファクトを生成 # mageモジュールのMAGESEクラスからSE用MAGEのインスタンスを作成 mage_img = mage.MAGESE(data) # シフトさせる最大のピクセル量 move_pixel = 10 move_range = np.arange(move_pixel*-1, move_pixel+1, 1) # 前後方向に動かすメソッドmove_vertical()を実行 mage_img.move_vertical(move_pixel) # 前後方向に動いた画像を表示 for k, _ in enumerate(move_range): plt.subplot(5, 5, k+1) plt.axis('off') plt.imshow(mage_img.vertical_moved_image[k], cmap='gray') plt.gca().title.set_text('Vertical move ('+str(k-10)+' pixel)')

擬似k-spaceの作成
次にシフトしたMRI画像をフーリエ変換することで擬似k-spaceに変換し、それらの複数の擬似k-spaceを位相エンコード方向にランダムサンプリングして、一枚のk-spaceを作成します。
私の実装では先ほどMRI画像をシフトさせたときに同時にk-spaceも作成し、またシフトした擬似k-spaceデータからランダムサンプリングして1枚のk-spaceを作成しています。そのため、あえてk-spaceを作成するコードを書く必要はありません。
モーションアーチファクトMRI画像の作成
シフトした画像をランダムにサンプリングしてモーションアーチファクトを模擬した擬似k-spaceを逆フーリエ変換します。これでモーションアーチファクトを生じさせたMRI画像が作成できます。
# 強度画像にして表示 img = np.abs(mage.kspace2img(mage_img.concate_vertical_moved_kspace)) plt.imshow(img, cmap='gray')

以上です。微妙にモーションアーチファクトが出ていると思います。
モーションアーチファクトの強度は調整できます。MAGEのコードの以下の部分のsdを大きくするほどアーチファクトが強く出るようになります。
def create_random_kspace(self, move_range, moved_kspace): concat_moved_kspace = np.zeros(self.matrix, dtype=np.complex64) # 乱数を設定 mean = np.median(move_range) sd = 5 random_number = self._get_trunked_normal(mean, sd, 0, np.max(move_range), self.matrix[self.pe]) random_number = np.ceil(random_number).astype(int)
TSEおけるMAGE
MAGEはSEとTSEの2種類のシークエンスでモーションアーチファクトを生成することができます。そこで私のpythonでの実装もMAGESEとMAGETSEの2つのclassを作成しました。どちらのシークエンスでもアーチファクト画像を作成できます。さきほどはSE版でアーチファクト画像を生成しました。TSE版では同じシフト量で以下のような画像が作成できました。
# TSE用のMAGEのインスタンスを作成 # 2つ目の引数はecho train length tse_img = mage.MAGETSE(data, 8) # シフトさせる最大のピクセル量 move_pixel = 10 # 前後方向に動かすメソッドmove_vertical()を実行 tse_img.move_vertical(move_pixel) # 強度画像にして表示 tse_mag = np.abs(mage.kspace2img(tse_img.concate_vertical_moved_kspace)) plt.imshow(tse_mag, cmap='gray')

論文の画像とはちょっと違うような気がしますが、なんとなくMAGEを再現できました。
ImageJのフーリエ変換を雑にマクロで紐解いてみる
私は画像のフーリエ変換をするときにImageJを使用することがあります.いちいちコードをかかずとも,マウスを数回クリックするだけで複雑な計算をしてくれるImageJはすばらしいソフトウェアだと思います.プログラムのコードを書くことは好きですが,画像一枚をフーリエ変換するようなときはGUIで行ったほうが効率的です.
ImageJのデフォルトの設定でフーリエ変換を行うと画像が一枚作成されるのですが,僕はこれが何なのか知りませんでした.よくない使い方ですが今まで雰囲気でなんとなくフーリエ変換を行っていました.以前の記事の最初に示したなんちゃってk-spaceもImageJのFFTを使用して作成しました.
知らないままにしておくのはまずいと思い,ImageJのデフォルト設定のFFTについて勉強しました.その結果,デフォルトのFFT後に現れる画像はパワースペクトルであり,画像としては不正確だが見た目を整えて人間が見やすい形にした画像であることがわかりました.表示されていないところで正確なデータが保持されており,計算をする際にはそちらの値を使用するそうです.そのためデフォルトのFFT後に表示される画像を保存したとしても,他の解析には使えないということがわかりました.
この記事ではImageJのFFTのデフォルト設定で行われている操作をマクロを使って雑に検証していきます.雑にというのはプログラムのソースコードを読めばわかるのに,僕はJavaが分からないので公式ドキュメントを参考にして自分なりに検証したからです.そのため計算が正確でないかもしれないし,どこか間違っている可能性もあります.その点をご了承ください.
目次
ImageJのFFTはFHTがベースとなっている
そもそもImageJのFFTは純粋な高速フーリエ変換ではないようです.ImageJ日本語情報のFFTの項目を見てみると以下の記載があります.
この機能は、NIHImage の 派生ソフト ImageFFT の作者である Arlo Reeves が製作した2次元の高速ハートレー変換(Fast Hartley Transform; FHT)を実装したものがベースになっている。
ImageJのFFTは高速ハートレー変換をベースにしているそうです.高速ハートレー変換なんてはじめて聞きました.
高速ハートレー変換(Fast Hartley Transform; FHT)
では高速ハートレー変換とはなんなのでしょう.日本語で以下の文献を見つけました.
この文献の緒言から高速ハートレー変換について簡単にまとめてみると
- フーリエ変換と極めて類似した変換である
- 実数の変換である
- 順・逆変換が等しい
といった特徴があるようです.(離散化)ハートレー変換,ハートレー逆変換を式で確認してみると


です.ここでcas(x)は

らしいです.
一方,(離散化)フーリエ変換,フーリエ逆変換は以下になります.


たしかに上で挙げたような特徴が式からうかがえます.よくわかりませんが,ImageJのFFTはハートレー変換がベースとなっているようです.
ImageJのFFT後の画像はパワースペクトル
公式ドキュメントを読み進めていくと,ImageJのFFT後の画像はパワースペクトルであるとの記載があります.さらに,このパワースペクトルは対数変換されており,8bitになっているようです.これらを理解するために,デフォルトのFFTで表示される画像(パワースペクトル)をマクロで作成していきます.
複素フーリエ変換
実数の2次元画像に対してフーリエ変換を行うと,複素数の2次元画像に変換されます.元画像f(n)をフーリエ変換して算出されるF(k)の右辺にはiが存在しているため,元画像f(n)が実数であっても複素数に変換されるということです.上のフーリエ変換の式のf(n)が画像でいうところの各画素値にあたり,F(k)がフーリエ変換後の各画素値になります.
複素数はそのままだと扱いづらいですが,実部と虚部に分けることで実数平面の2つの画像として表示することができます.まずは画像をフーリエ変換し,実部と虚部に分けて表示させます.ImageJを開きctrl+shift+Nを同時に入力,その後表示されたText Windowに以下のマクロを入力し,ctrl+Rで実行します.
run("T1 Head (2.4M, 16-bits)");
setSlice(64);
run("FFT Options...", "complex do");
このようにすることでImageJのサンプル画像(T1WI head sagital)を表示し,そのフーリエ変換後の画像を実部と虚部に分けて表示することができます.

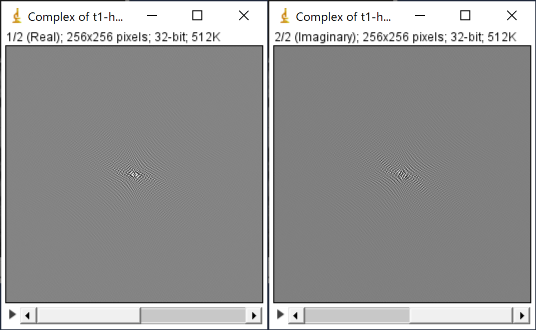
ここでImageJのFFTはFHTベースと書きましたが,なぜ複素数の実部と虚部の値が出て来るのかが不思議だったのですが,wikipediaを見てみますと正規化の係数として1/21/2を用いる時のフーリエ変換とハートレー変換は以下の式で表されるそうです.

パワースペクトル
次にこれらの実部と虚部の画像から理論上のパワースペクトルを作成します.パワースペクトルはF(k)の絶対値の二乗であらわされます.つまり実部と虚部の二乗和になります.マクロは以下の通りです.
selectWindow("Complex of t1-head.tif");
run("Square", "stack");
run("Stack to Images");
imageCalculator("Add create 32-bit", "Real","Imaginary");
なおここで作成した理論上のパワースペクトルはFFT OptionsのなかのRaw Power Spectrumを選択すれば作成可能です.手動で作成したものと,Raw Power Spectrumで作戦される画像は一致していました.

対数変換と8bit画像への変換
ImageJのデフォルトのFFTではこの理論上のパワースペクトルの対数を取り,最小値が1、最大値が254になるようになんらかの処理を行なっています.そして32bit画像であったパワースペクトルを8bit画像に変換しているようです.
この処理が公式ドキュメントを読むだけではいまいち理解できませんでした.仕方ないので読めないソースコードをなんとなく雰囲気で読み,こちらの質問も参考にしてみました.
//対数変換
selectWindow("Result of Real");
run("Log");
//(1~254に)線形変換
//この画像の最大値が30.413、最小値が6.18
run("Macro...", "code=v=(v-6.18)/(30.413-6.18)*(254-1)+1");
//8bitに変換
run("Conversions...", " ");
run("8-bit");

デフォルトのFFT後のパワースペクトルと比較
デフォルトのFFTで作成された画像とヒストグラムを表示し,比較してみます.
//ImageJのデフォルト設定のFFTを実行
selectWindow("t1-head.tif");
run("FFT Options...", "fft do");
//それぞれのヒストグラムを作成
selectWindow("Result of Real");
run("Histogram", "bins=256 x_min=0 x_max=255");
selectWindow("FFT of t1-head.tif");
run("Histogram", "bins=256 x_min=0 x_max=255");


ヒストグラムは左側が手動,右側がデフォルトのパワースペクトルのものです.デフォルトのフーリエ変換後の画像は手動で計算した画像と類似しており,ヒストグラムの形状も同様に類似していますが,値は同じになりませんでした.どこかで計算間違いをしているからだと思いますが,原因はわかりません.誰か教えてください.
まとめ
デフォルトのパワースペクトルを手動で(なんとなく)計算できました.ImageJのデフォルトのFFTがいろいろな処理をしていることがわかりました.
なぜImageJのFFTがこのような仕様なのかを自分なりに考察してみました.まず,FFTをパワースペクトルで表示することについてですが,パワースペクトルが最も適した形式であったからだと思いました.そもそもフーリエ変換は周波数解析をするために行われるので,各周波数の成分の大きさを観察するためにはパワースペクトルで表示するのが良いはずです.また,複素数を2次元画像として表現するのが困難なことも理由の一つだと思います.
ちなみにパワースペクトルがパワーと呼ばれる所以を調べてみたところ,以下の説明を見つけました.なんとなくわかった気になれます.
物理学のいろいろな問題で振動のエネルギーが1/2(定数)×(振幅)2で表されます.この定数は問題によって,物体の質量であったりコンデンサーの静電容量(キャパシタンス)であったりコイルの誘導係数(インダクタンス)であったりします.ですから,振幅の二乗をエネルギーとみなすのは自然です.しかし,ここでは物理学でいうエネルギーというより,形式的に考えた”エネルギーのようなもの”と考えてください.(金谷健一(2003)『これなら分かる応用数学教室 最小二乗法からウェーブレットまで』共立出版,p.99)
その他の処理についてですが,対数を取るのは小さな値を底上げする(観察しやすくする)ためであり,線形変換をして8bitにするのは人間の目が8bit程度しか認識できないからそうしているのだと思います.
つまり,ImageJのデフォルトのFFTはわざわざ丁寧に人間が目で見やすいような形に調整してくれているのだと思います.
まとまりがないですが,最後に今回使ったImageJのマクロを以下にまとめて終わりにしたいと思います.
//サンプル画像を読み込み
run("T1 Head (2.4M, 16-bits)");
setSlice(64);
//(複素)フーリエ変換
run("FFT Options...", "complex do");
//パワースペクトルの算出
selectWindow("Complex of t1-head.tif");
run("Square", "stack");
run("Stack to Images");
imageCalculator("Add create 32-bit", "Real","Imaginary");
//ImageJのデフォルト設定のFFTから算出される画像に寄せる
selectWindow("Result of Real");
//対数変換
run("Log");
//(1~254に)線形変換
//この画像の最大値が30.413、最小値が6.18
run("Macro...", "code=v=(v-6.18)/(30.413-6.18)*(254-1)+1");
//8bitに変換
run("Conversions...", " ");
run("8-bit");
//デフォルトのパワースペクトルと作成したパワースペクトルのヒストグラムを比較
//ImageJのデフォルト設定のFFTを実行
selectWindow("t1-head.tif");
run("FFT Options...", "fft do");
//それぞれのヒストグラムを作成
selectWindow("Result of Real");
run("Histogram", "bins=256 x_min=0 x_max=255");
selectWindow("FFT of t1-head.tif");
run("Histogram", "bins=256 x_min=0 x_max=255");
````
MRI画像の画素値がしたがう確率分布をPythonで理解する
以下の有名な論文があります.
この論文によればMRI画像は
- 正規分布
- Rician (Rice) 分布
- Rayleigh分布
の3つのいずれかの確率分布にしたがうとされています.簡単に言えば,SNRが良いピクセルは正規分布,悪いピクセルはRice分布,信号がない箇所(バックグラウンド)はRayleigh分布にしたがうようです.
今回の記事では,MRI画像にノイズを付加した画像を作成し,上述した確率分布が得られるのかを検証しました.MRI画像はエムアールアイシミュレーションズさんのBlochSolverで作成した画像を用いています.
また,今回検証に使用した画像は,以下の以前の記事で作成したファントムデータのT2WIを使用しています.
ノイズを付加した画像の作成
MRI画像はなんらかのノイズの影響を受けているため,確率分布にしたがってピクセル値は変動していると考えられています.ノイズの原因はさまざまですが,今回とりあげるのは熱雑音です.以下にwikipediaの熱雑音の説明を載せています.
熱雑音(ねつざつおん、英: thermal noise)は、抵抗体内の自由電子の不規則な熱振動(ブラウン運動[1])によって生じる雑音のことをいう。
MRIの場合,熱雑音はコイルなどから発生していると考えられます.また,熱雑音は正規分布に従うと仮定されることが多いようです.
以前の記事で取り上げたように,MRIは直角位相検波という手法でreal信号とimaginary信号を取得しています.そのため,real信号とimaginary信号に対して,正規分布にしたがうノイズを付加することで,実際のMRI画像と同様のノイズを付加したことを模擬できます.以下,そのコードです.
import numpy as np import matplotlib.pyplot as plt from PIL import Image path = 'result/2020-11-26/2D_SpinEcho-15-complex.flt' matrix = 256 # numpyでバイナリデータを読みこむ def bin2kspc(path, matrix): raw = np.fromfile(path, dtype='f', sep='') real = raw[::2].reshape(matrix, matrix) imag = raw[1::2].reshape(matrix, matrix) return real, imag # real imageとimaginary imageに正規分布にしたがうノイズを付与 def add_noise(real, imag, sd): shape = np.shape(real) noise_r = np.random.normal(0, sd, shape) noise_i = np.random.normal(0, sd, shape) real += noise_r imag += noise_i return real, imag # realとimaginaryを複素数としてひとまとめにする def make_comp(real, imag): real = real.astype(np.complex64) imag = imag.astype(np.complex64) imag *= 1j kspace = real + imag return kspace # 逆フーリエ変換 def ifft(kspace): ifft = np.fft.ifft2(kspace) ifft = np.fft.ifftshift(ifft) real_ifft = ifft.real imag_ifft = ifft.imag abs_ifft = np.abs(ifft) return real_ifft, imag_ifft, abs_ifft def main(sd): real, imag = bin2kspc(path, matrix) n_real, n_imag = add_noise(real, imag, sd) kspace = make_comp(n_real, n_imag) real, imag, magn = ifft(kspace) return real, imag, magn # sd=10とsd=50のノイズを付加したで画像を作成 real_sd10, imag_sd10, magn_sd10 = main(sd=10) real_sd50, imag_sd50, magn_sd50 = main(sd=50) # データを表示 fig = plt.figure(figsize=(7,4)) plt.axis('off') ax1 = fig.add_subplot(1,2,1) ax2 = fig.add_subplot(1,2,2) ax1.imshow(magn_sd10, cmap='gray',vmin=0,vmax=1) ax1.set_title('Magnitude (sd=10)') ax1.axis("off") ax2.imshow(magn_sd50, cmap='gray',vmin=0,vmax=1) ax2.set_title('Magnitude (sd=50)') ax2.axis("off") plt.savefig('magnitude.png')

k-space上に標準偏差が10と50の正規分布にしたがうノイズを加えたMRI画像を作成しました.sd=50の画像はかなりノイジーに見えます.
大量のデータの作成
次に同じsdの正規分布にしたがうノイズを発生させ,そのノイズを付加した画像を複数枚作成します.こうすることで,擬似的に複数回ファントムを撮像した状態を模擬できます.今回知りたいのはMRIの画素値がどのような確率分布にしたがうのかということなので,シミュレーションによりノイズが付加された画像を大量に作成します.
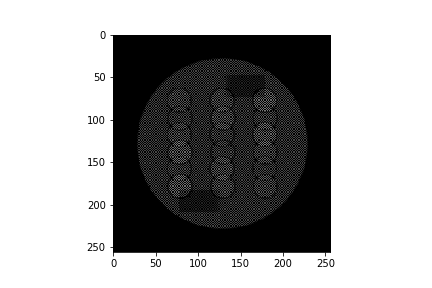
画像を作成した後,すべての画像上の左上(バックグラウンド)と画像中の3×3の丸がある中の2行目のすべての丸のなかの画素値をひとつ取得します.つまり4つのROI (1pixel) を設定します.下の画像のA,B,C,Dの位置です.

import pandas as pd from tqdm import tqdm images = {'sd10':[],'sd50':[]} d = {'magn_sd10_A':[], 'magn_sd10_B':[], 'magn_sd10_C':[], 'magn_sd10_D':[], 'magn_sd50_A':[], 'magn_sd50_B':[], 'magn_sd50_C':[], 'magn_sd50_D':[]} for j in tqdm(range(5000)): images['sd10'].append(main(sd=10)[2]) images['sd50'].append(main(sd=50)[2]) for i in tqdm(range(5000)): # A = [0,0], B = [80,140], C = [130,140], D = [180,140] d['magn_sd10_A'].append(images['sd10'][i][0,0]) d['magn_sd10_B'].append(images['sd10'][i][80,140]) d['magn_sd10_C'].append(images['sd10'][i][130,140]) d['magn_sd10_D'].append(images['sd10'][i][180,140]) d['magn_sd50_A'].append(images['sd50'][i][0,0]) d['magn_sd50_B'].append(images['sd50'][i][80,140]) d['magn_sd50_C'].append(images['sd50'][i][130,140]) d['magn_sd50_D'].append(images['sd50'][i][180,140]) df = pd.DataFrame.from_dict(d)
100%|██████████| 5000/5000 [00:47<00:00, 104.65it/s]
100%|██████████| 5000/5000 [00:00<00:00, 29689.48it/s]
5000回撮像したファントム画像のA,B,C,Dの位置の画素値のデータを取得しました.
次にfitterというPythonのライブラリを使って,それぞれのピクセルがどの確率分布にしたがうのか検証します.fitterは最尤法を用いて,ピクセルの集合のヒストグラムから当てはまりの良い確率分布を探し出すことができるライブラリです.
from fitter import Fitter result = {'magn_sd10_A':[], 'magn_sd10_B':[], 'magn_sd10_C':[], 'magn_sd10_D':[], 'magn_sd50_A':[], 'magn_sd50_B':[], 'magn_sd50_C':[], 'magn_sd50_D':[]} for i in result.keys(): result[i] = Fitter(df[i], distributions=['norm', 'rice', 'rayleigh'])
以上でsd=10とsd=50のノイズを加えた画像のA,B,C,Dの位置のピクセルがしたがう確率分布の結果を計算できました.
それではMRI画像がどのような確率分布にしたがうのかを見ていきます.まずはAの位置(バックグラウンド)のsd=10とsd=50のバックグラウンドのピクセルがしたがう確率分布を見ていきます.
# sd=10 result['magn_sd10_A'].fit() result['magn_sd10_A'].summary()
| sumsquare_error | aic | bic | kl_div | |
|---|---|---|---|---|
| rayleigh | 70.486597 | -137.259822 | -21291.818686 | inf |
| rice | 70.968561 | -130.667313 | -21249.229462 | inf |
| norm | 283.554135 | -23.668768 | -14331.916283 | inf |

# sd=50 result['magn_sd50_A'].fit() result['magn_sd50_A'].summary()
| sumsquare_error | aic | bic | kl_div | |
|---|---|---|---|---|
| rice | 2.401464 | 129.260283 | -38180.021089 | inf |
| rayleigh | 2.406282 | 124.249910 | -38178.517287 | inf |
| norm | 10.665122 | 209.537389 | -30734.037491 | inf |

表は左列から二乗和誤差,AIC,BIC,カルバック・ライブラー情報量を示しています.どの指標も値が小さいほど良いことを示します.グラフはヒストグラムがデータの分布で,曲線はデータを確率分布にフィッティングさせたときに求めたパラメータで作成された確率分布を示しています.
sd=10ではRayleigh分布の二乗和誤差がもっとも低値でしたが,sd=50ではRice分布の二乗和誤差がもっとも低値を示しました.よく見てみると,sd=50ではAICとBICはRayleigh分布の方が低値を示しているので,実質的にどちらのsdの画像もRayleigh分布にしたがっていると考えることができそうです.
次にBの位置を見ていきます.
# sd=10 result['magn_sd10_B'].fit() result['magn_sd10_B'].summary()
| sumsquare_error | aic | bic | kl_div | |
|---|---|---|---|---|
| norm | 20.220786 | -7.820432 | -27535.376053 | inf |
| rice | 20.230288 | -5.692187 | -27524.509975 | inf |
| rayleigh | 770.452108 | -203.635577 | -9334.044093 | inf |

# sd=50 result['magn_sd50_B'].fit() result['magn_sd50_B'].summary()
| sumsquare_error | aic | bic | kl_div | |
|---|---|---|---|---|
| norm | 1.450872 | 235.523199 | -40708.108874 | inf |
| rice | 1.451824 | 228.592473 | -40696.311037 | inf |
| rayleigh | 16.732950 | 115.898379 | -28482.032280 | inf |

こちらは表の値とヒストグラムをみた感じでは,どちらも正規分布にしたがっていると考えて良さそうです.
次にCの位置です.
# sd=10 result['magn_sd10_C'].fit() result['magn_sd10_C'].summary()
| sumsquare_error | aic | bic | kl_div | |
|---|---|---|---|---|
| rayleigh | 70.767290 | -194.946403 | -21271.947146 | inf |
| rice | 70.774084 | -192.916473 | -21262.949908 | inf |
| norm | 274.531438 | -109.723563 | -14493.602664 | inf |

# sd=50 result['magn_sd50_C'].fit() result['magn_sd50_C'].summary()
| sumsquare_error | aic | bic | kl_div | |
|---|---|---|---|---|
| rayleigh | 3.147475 | 139.233666 | -36835.929129 | inf |
| rice | 3.147479 | 141.245974 | -36827.405657 | inf |
| norm | 13.302968 | 226.980239 | -29628.995801 | inf |

Cの位置では,どちらのsdの画像もRayleigh分布もしくはRice分布にしたがっているように見えます.なお,DはBほぼ同様の結果になったので省略します.
まとめ
バックグラウンド領域はRayleigh分布に,信号発生領域では正規分布もしくはRice分布にしたがっていることが確認できました.最初にあげた論文では,バックグラウンド領域はRayleigh分布にしたがうとありましたので,その通りになっていることが確認できました.
気になるのは信号発生領域はどのようなときに正規分布もしくはRice分布にしたがうのかということです.論文中ではSNR<2のピクセルはRice分布,SNR>2のピクセルは正規分布にしたがうとありました.そこで今回のBとCのSNRを算出してみます.
def SNR(d): return np.mean(d)/np.std(d) print(f"SNR of B (sd=10): {SNR(df['magn_sd10_B'])}\nSNR of B (sd=50): {SNR(df['magn_sd50_B'])}\nSNR of C (sd=10): {SNR(df['magn_sd10_C'])}\nSNR of C (sd=50): {SNR(df['magn_sd50_C'])}")
SNR of B (sd=10): 12.391050217474877
SNR of B (sd=50): 2.8108878515892477
SNR of C (sd=10): 1.9194703847218806
SNR of C (sd=50): 1.9089855205754447
BはどちらのsdにおいてもSNR>2でしたが,CはどちらのsdにおいてもSNR<2でした.論文と整合性がとれてることが確認できました.
参考にした記事
「対話形式だからわかりやすい!MRIの教科書」は初めてのMRIの教科書としてオススメ
本記事は「対話形式だからわかりやすい!MRIの教科書」の第1章と第2章の書評記事です。 本書は難しい数式を使わず噛み砕いた表現を使うことで、MRI の原理を気軽にわかりやすく理解することを目的に企画されたようです。一貫して対話形式で進む登場人物たちのやり取りを読むことで、難しいMRIの基礎原理を習得することができます。そのため本書はこれからMRIを学びたいと考えている人にとっての「最初の1冊」として適した本であると思います。
目次
きっかけ
ちょっと前にMRIの情報収集をしているときに面白そうなタイトルの本書を見つけました。タイトルを見て中身を読んでみたいと思いましたが、買おうとまでは思えませんでした。著者情報の記載はあるも、誰だかわからないし、個人出版のようなので情報に信頼性があるかがわからなかったからです。
そんなときに以下のツイートを見ました。Kindleストアで著者のTwitterアカウント(@MRI_taiwa)が記載されていたので、フォローしていたので気づくことができました。
どなたか、私の書籍「MRIの教科書」を広めるお手伝いをしてくださいませんか?https://t.co/efA0D3Nw1N
— MRIの教科書@対話形式だからわかりやすい! (@MRI_taiwa) 2020年8月7日
草案かつ応相談ですが、
・対象書籍は第1章、第2章
・契約は月単位(直近だと9月1日〜30日)
・該当1か月分の印税収入をお支払い
(Amazonポイント1000円単位)
のような条件を考えています。
このツイートに乗っかれば、気になってた本が読める!と思い、ツイートに返信してみました。数回のやり取りの後、この記事の執筆にいたりました。つまり、ブログでこちらの本を宣伝する代わりに本を読ませていただく(報酬を頂く)こととなりました。
可能な限りニュートラルな立場で書評をしたつもりですが、この点を理解した上で読んでいただければと思います。それでは以下に書評の内容を書いていきます。
本書はMRI基礎原理を学ぶために企画されている
この本の対象はMRIの基礎原理について興味がある、しかし、これまでじっくりと取り組んだことのない方、あるいは一度取り組もうとしたけれど挫折をした方だそうです。たしかに本書の内容はMRIの基礎について広く、浅く、易しく書かれています。
第1章はMRI検査全体についての大まかな説明
第1章を読むとMRIについての全体像を把握することができます。MRIの原理は非常に難しいため、まずは全体を知って、それから細かなところを深堀するほうがいいという筆者の考えから、この章を設けたようです。MRIのことをよく知らない人でも1章に書いてあることはなんとなく聞いたことがあると思います。
第2章では本格的にMRI装置のことについて
第2章ではMRI装置本体の原理を知ることができます。MRIは複数のコイルから成り立つという話から始まり、その後はそれぞれのコイルの役割について詳しく説明されています。
第2章は図をふんだんに使われており、筆者の熱量を感じられます。傾斜磁場の説明ではかなり頑張って作図されているということが垣間見えます。わかりやすく説明するために工夫を凝らしてある、素晴らしい本だと思いました。
本書の良い点
- 対話形式は内容が頭に入ってきやすい
- 動画(GIF)が面白い
本書はAさんとBさんと博士の3人の対話で内容が進んでいきます。僕が知る限り対話形式のMRIの本はありません。あえていうとすれば、荒木力先生の「MRI完全解説」をストーリーにしたものといった感じでしょうか。「MRI完全解説」はQ and A方式の教科書であり、趣向としては似ているのかなと思いました。ちなみに「MRI完全解説」は僕が磁気共鳴専門技術者試験に合格するために、最も参考にした書籍です。
また、本書は電子書籍の特徴を活かし、要所要所に動画(GIF)のリンクが貼られています。
gifアニメを作ってみた #MRI,#kindle_publishing pic.twitter.com/bcQuMju4ON
— MRIの教科書@対話形式だからわかりやすい! (@MRI_taiwa) 2020年4月28日
これは非常に面白い試みだと思いました。MRIの原理は静止画だと理解が難しいため、動画で見ることができればその一助になると思います。これからもGIFをたくさん作っていただけるとさらに面白い本になると思いました。
本書の惜しい点
- 登場人物がわからなくなる
- 章ごとで販売されているため、1冊で考えたときの値段が高い
本書はAさんとBさんと博士の3人のやりとりで話が進んでいきます。僕は頭のメモリが弱いので、たびたびAさんとBさんがどっちがどっちかわからなくなりました。名前で表記していただければそのストレスもなかったのかなと思いました。
そして本書の値段ですが1章が300円、2章が500円となります。たしかにそれぞれの章で話は完結していますが、すべての章を合わせた1冊の値段で考えるとそれなりの値段になります。まとめ買いをすれば安く買える仕組みがあってもいいかと思いました。
しかしながら、本書はkindle unlimitedに登録すれば無料で読めます。30日間無料体験キャンペーンを長いことしているようです。本書が気になるけど、お金を出したくない方はkindle unlimitedも一つの方法かと思います。
今後の章に期待大!
2020年8月現在、まだ2章分しか販売されていませんが、その内容はMRIを学ぼうと考える人にとっての「初めの1冊」として非常に良いものだと思います。
とはいうものの、僕は本書に記載されていた「シールドフィンガー」や「シム」の言葉の由来について全く知りませんでした。臨床で直結する知識ではなく、いわゆる豆知識ですが、こういったセレンディピティともいえる発見があったので、結果的に僕は読んでよかったと思いました。
今後も続編発刊が予定されているようですので、続編にも大きく期待したいと思います。
Pythonを使ってk-spaceを理解する[フーリエ変換編]
k-spaceはよくこんな感じで説明されてます.

k-spaceはMRIで受信した信号を格納したものと私は理解してます.k-spaceを逆フーリエ変換するとMRI画像が取得できます.
ところで,大手医療機器メーカーが販売しているMRI装置では,ユーザーは基本的にk-spaceを見ることはできません.生データが格納されているので,ユーザーに知られたくない情報が明らかになってしまう恐れがあるからだと私は思ってます(各社メーカー独自のフィルタ等の処理がされている?).そのため,我々診療放射線技師はk-spaceの存在を知ってはいるものの,実際に見る機会は少ないです.k-spaceはアーチファクトやSNRや分解能を理解するのに不可欠なものです.もしそれを直接触ることができたら,MRIについてもっと理解が深まると思いますが,簡単にはできませんでした.
しかし,今はできるようになりました.前回紹介した巨瀬先生のMRIシミュレータ本ではk-spaceを取得することができます.
前回はBlochSolverの簡単な使い方を試したのですが,今回はMRIシミュレータで作成したk-spaceをPythonを使ってフーリエ変換を行い,MRI画像を再構成してみました.装置がやることを自分で実際にやってみて理解を深めることが目的です.
k-spaceは2つ存在する
そもそもMRIは信号を直角位相感受性検波という手法を用いて取得しています.基準周波数と90度位相のずれた基準周波数の両方の信号を受信する方法です.この方法を用いることで正確に信号を認識できるらしいです.また,基準周波数の信号を実信号,90度ずれた信号を虚信号と言います.つまり,2種類の信号のデータが存在します.ということはk-spaceは実信号と虚信号の2つ分存在すると考えることができます.研究会や学会で演者の人が見せるスライドには,この記事の最初に示したような1つのk-spaceで説明されることが多い印象ですが,正確には2つデータがあるので実信号のk-spaceと虚信号のk-spaceの2つを考える必要があります.
実際にk-spaceを見てみる
ここからMRIをシミュレーションしていきます.シーケンスはスピンエコーを使用しました.シミュレーションの対象はスライス評価ファントムです.以下その画像.

Matrix = 256×256,TR/TE = 4000/100 msecでシミュレーションしました.シミュレーションのやり方は前回の記事を参考にしてください.
さて,シミュレーションで作成されたデータはResultフォルタ内にシーケンス名-complex.fltという形で保存されています.このデータは,MRI信号の実信号と虚信号が32bit浮動小数点で交互に保存されているようです.まずはPythonを使って交互に保存されたデータをそのまま見てみます.
import numpy as np import matplotlib.pyplot as plt from PIL import Image # データが存在するパスを指定 data = 'シーケンス名-complex.flt' # データを読み込む with open(data, 'rb') as f: d = f.read() # バイナリデータをイメージ形式に変換 raw = Image.frombytes('F', (512,256), d) # 画像を保存 plt.imshow(raw) plt.savefig("raw.png")

次に,このデータを実信号と虚信号に分けてみます.
# 要素が0の入れ物を作っておく real = np.zeros([256,256]) imag = np.zeros([256,256]) # データを分割 for i in range(256): for j in range(256): real[i,j] = raw[i,2*j] imag[i,j] = raw[i,2*j+1] # それぞれを保存 fig = plt.figure() ax1 = fig.add_subplot(1,2,1) ax2 = fig.add_subplot(1,2,2) ax1.imshow(real, cmap='gray') ax1.set_title('Real') ax2.imshow(imag, cmap='gray') ax2.set_title('Imaginary') plt.savefig('real_imag.png')

フーリエ変換
ここから診療放射線技師養成校で習うものの中でも鬼門となるフーリエ変換のパートに入ります.実信号と虚信号のデータを取得できたので,これらを使って絶対値表現されたMRI画像を作ります.
ここで私は今までたいへんな勘違いをしていることに気づきました.通常用いるいわゆるMRI画像は,実画像と虚画像との絶対値画像であるとは知っていましたが,実画像は実信号が入ったk-space,虚画像は虚信号が入ったk-spaceをそれぞれフーリエ変換して,それらの絶対値をとって作成するものだと思ってましたが,違いました.実際に実信号だけで実画像を作成してみます.
# ダメな例 bad_real = np.fft.ifft2(real) bad_real = np.fft.ifftshift(bad_real) bad_real = bad_real.real plt.imshow(bad_real, cmap='gray') plt.savefig('bad_real.png')
実際にやってみてびっくり.実信号のみでフーリエ変換を行うとひっくり返った画像が重なってしまいます.
このことについて,金沢工業大学の樋口先生のホームページにわかりやすい説明がありました.
以下樋口先生のホームページからの引用です.
実k空間信号のみで画像再構成を行った場合、先に述べたように負の周波数成分があるため2つの画像が出現する。本例ではその一部が重なりあっている。重なりをなくすためにはk空間信号の最高周波数を下げる、すなわち空間分解能を下げる必要がある。これに対して複素k空間信号による再構成画像は負の周波数成分が発生しないため画像は1つのみである。したがって、複素k空間信号の方がフーリエ空間上の点を最大限利用できる。
こんなの知らなかった…でも,シミュレーションでk-spaceを触れたからこそ,新たに知ることができました.
話を戻して,ではどのようにすれば実画像と虚画像が作成できるかというと,樋口先生が記載しているように実信号と虚信号を複素数にして1つにまとめ,それをフーリエ変換すればいいようです.つまり,k-spaceを以下のように複素数で表現します.
ここでが実信号のk-spaceのデータ,
が虚信号のk-spaceのデータです.
次に簡単のため,1次元の逆フーリエ変換の式を以下に示します.
またオイラーの公式は以下の通り
よって最初の逆フーリエ変換の式は以下のように変形できます.
このとき,実画像と虚画像は以下のように表されます.
これらを書き直すと,
となります.ここでが実画像,
が虚画像になります.
そして通常よく用いられる絶対値MRI画像は以下の式で求められます.
実信号のk-spaceと虚信号のk-spaceの2つを複素数として結合して,逆フーリエ変換をすれば,実画像,虚画像が複素数として表現された正しい値が取得できるようです.この式変形は下に書いてある参考文献の『Excelによる医用画像処理入門』を参考にしました.どこか間違ってたら教えていただければありがたいです.
ということでこれをPythonでやってみます.
# 複素数の配列を作って実信号と虚信号を1つの複素数とする com = np.zeros([256,256], dtype=np.complex64) for i in range(256): for j in range(256): com[i,j] = complex(real[i,j],imag[i,j]) # 複素数データを逆フーリエ変換をする ifft = np.fft.ifft2(com) ifft = np.fft.ifftshift(ifft) # 実画像,虚画像,絶対値画像に分ける real_ifft = ifft.real imag_ifft = ifft.imag abs_ifft = np.abs(ifft) # データを表示 fig = plt.figure(figsize=(8.0, 4.0)) ax1 = fig.add_subplot(1,3,1) ax2 = fig.add_subplot(1,3,2) ax3 = fig.add_subplot(1,3,3) ax1.imshow(real_ifft, cmap='gray',vmin=0,vmax=1) ax1.set_title('Real') ax2.imshow(imag_ifft, cmap='gray',vmin=0,vmax=1) ax2.set_title('Imaginary') ax3.imshow(abs_ifft, cmap='gray') ax3.set_title('Magnitude') plt.savefig('final_data.png', )
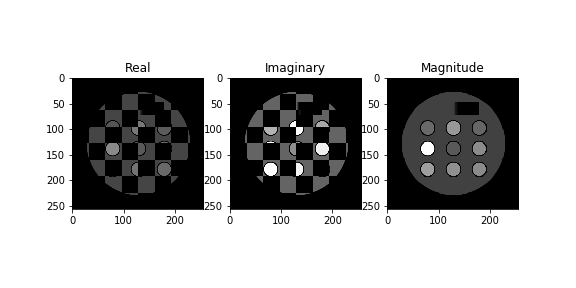
こうするとなぜか実画像と虚画像に縞模様が・・・.別々で保存してみます.
# real plt.imshow(real_ifft, cmap='gray',vmin=0,vmax=1) plt.title('Real') plt.savefig('real_ifft.png') # imag plt.imshow(imag_ifft, cmap='gray',vmin=0,vmax=1) plt.title('Imaginary') plt.savefig('imag_ifft.png')


縞模様が消えました.なぜ縞模様がでるのだ…とりあえず絶対値画像が作れたのでよしとします.
まとめ
k-spaceってほんと難しい.直接見れないのもあるし,フーリエ変換とかいうよくわからない強敵もいるし,なにかと理解を拒む壁があります.でもシミュレーションデータで仮想的にk-spaceを作って自分で再構成をすることで,少し理解できた気になれました.まだまだ知らないことだらけです.次はもう少しフーリエ変換について勉強して記事を書こうと思います.そのあとはk-spaceをいろいろいじってMR画像にどのような影響が出るかシミュレーションしてみたいと思います.
参考文献

- 作者:荒木 力
- 発売日: 2014/03/27
- メディア: 単行本
被ばく線量情報抽出ソフト『DoNuTS』を作りました

2020年4月1日より医療法施行規則の一部を改正する省令が施行され,CTなど一部の放射線科領域の医療機器を有する施設は被ばく線量の記録・管理が義務付けられました.多くの大規模施設では線量管理ソフトと呼ばれる高価なソフトを導入し,PACSと紐付けるなどして,簡便に線量の管理を行えるような体制を整えつつあります.しかし,大規模施設と比べて放射線科の検査件数が少ない中小規模施設では,新たに線量管理ソフトを導入することは困難であると考えられます.
そこでRDSR(Radiation Dose Structured Report)とPET画像のヘッダーからCTとPET/CTの被ばく線量情報を抽出し,csvで出力できるフリーソフト『DoNuTS(Dose Counter for Nuclear Medicine and CT Systems)』を作りました.DoNuTSを使用することで,線量管理ソフトの導入が困難な施設においても,線量管理を簡便に行うことができると考えられます.今回はDoNuTSの紹介をしたいと思います.
なお,ソースコードは公開しており,無料でお使いいただけます.ただし,努力して作りましたがプログラミングに関しては素人が作ったため,バクがある可能性があります.ご使用は自己責任でお願いいたしますm(__)m
作成した方法
使用したプログラミング言語はPython(version 3.6.9)です.windows上でソフトとして使用する為には.pyファイルを.exeファイルにする必要があります.今回はpyinstallerを使用してexe化しました(2021.1.30追記 macでも動作するようになりました).
ダウンロードの方法
ソフトは以下のリンクからダウンロードできます.
リンクをクリックすると,僕のGithubのページに飛びます.
右上のあたりにある緑色のClone or downloadをクリックすると,ファイルのダウンロードが始まります.ダウンロードしたファイルの中にsrcという名前のフォルダがあります.そのsrcの中にdistというフォルダがあり,その中にDoNuTS.exeというファイルがあります.それがDoNuTSの本体となります.
2021.1.30 追記
Githubのアップデートにより,webページのレイアウトが以下のように変わっています.
DoNuTS本体のダウンロード方法も変更しています.右上あたりのRelease v0.1.0をクリック.zipファイルをダウンロードし,解凍すれば実行ファイル(.exeまたは.app)があり,それを使ってください.(追記終わり)
使用方法
データの準備
まずはRDSRとPET画像をDoNuTSを動作させるPC上にexportしておく必要があります.このとき,1つのフォルダ内にデータを取得したいRDSRおよびPET画像を保存してください.なお,DoNuTSはRDSRとPET画像のみを探して処理を行うため,他のファイルがフォルダ内に存在していても動作に問題はありません.
DoNuTSの実行
データの準備ができたらDoNuTSをダブルクリックして実行します.するとコマンドプロンプトが立ち上がります.黒い画面のアレです.次に,取得したいデータが入ってあるフォルダを指定するよう指示されるので,指示に従い先ほど準備したフォルダを選択します.処理が終わると以下の図のようなポップアップが表示されます.
これで処理は完了です.なお,出力されるデータは
- Mean CTDIvol
- DLP
- X-Ray Modulation Type
- CTDIw Phantom Type
- Acquisition Protocol
- Target Region
- CT Acquisition Type
- Procedure Context
- Exposure Time
- Scanning Length
- Exposed Range
- Nominal Single Collimation Width
- Nominal Total Collimation Width
- Pitch Factor
- Identification of the X-Ray Source
- KVP
- Maximum X-Ray Tube Current
- Mean X-Ray Tube Current
- Exposure Time per Rotation
- Device Manufacturer
- Device Serial Number
- Manufacturer Model Name
- Patient ID
- Study Date
- Patient Name
- Study Description
- Patient BirthDate
- Patient Sex
- Patient Age
- Patient Size
- Patient Weight
- CT Dose Length Product Total
- Radionuclide Total Dose (PET画像が存在した場合)
2020.7.17 追記
(出力できるデータが増えたため,リストを変更しました.)
以上です.出力されたcsvは,先ほど指定したフォルダ内に保存されています.
実際の業務における使用例
診療放射線技師会や医学放射線学会のホームーページで線量管理に関するガイドラインが発表されています.その中身を確認してみるとおおよそ以下のことをしておけば問題はないと考えられます.
- 線量の記録
- dose summaryをPACSに転送しておく
- 線量の管理
- 記録してある線量情報をサンプリングし,自施設の線量を算出
- DRL等を利用して自施設の線量を評価し,最適化を行う
ここでDoNuTSは線量の管理で効果を発揮します.たとえばCTの線量管理の場合,検査終了後に撮影した患者のRDSRをPACS等に保存する運用をしていき,線量の管理を行う際に保存しておいたRDSRをexportし,DoNuTSを使用します.そうすることでDLPや体重が患者ごとにcsvとして出力されるので,Excel等をうまく利用すれば被ばく線量の最適化を簡便に行うことができると考えられます.
まとめ
最近の装置はRDSRを出力できるのが一般的になっています.RDSRは構造化されたレポートという名前の通り,データを取り出すことさえできれば,非常に便利なファイルです.しかし,実際は高価な線量管理ソフトを導入しなければ,RDSRからデータを取り出すこと自体困難です.DoNuTSはRDSRの中の線量管理に必要な情報を簡便に取り出すことができるので有用だと思います.是非使ってみて下さい.
また,ソフトに関する要望やバグを発見した場合は,Github上のissueに書き込むか,メール(sugimotokouhei(at)gmail.com)をしていただくか,なんらかの手を使って僕にお知らせいただければ幸いです.