Pythonのパッケージ「dicom2nifti」を使ってDICOMをNIfTIに変換する
NIfTIとは
医用画像のファイルフォーマットは基本的にDICOM形式ですが、DTIやfMRIなどのいわゆる脳のアドバンスドな解析に用いられるデータはNIfTI (Neuroimaging Informatics Technology Initiative) 形式に変換して解析することが一般的なようです。「.nii」や「.nii.gz」といった拡張子がNIfTI形式に相当します。「.nii.gz」は「.nii」をgzip圧縮したフォーマットです。
DICOMはスライスごとにファイルが作成されますが、NIfTIはひとつのシーケンスごとのすべてのスライスの画像をひとつのファイルとして保存することができます。この点がNIfTIの最大のメリットかと思います。ちなみにDICOMのヘッダーはNIfTIにおいてもヘッダーとして一部保持されます。
DICOMをNIfTIに変換するためには専用のソフトウェアが必要です。調べてみたところ、先人たちがさまざまなソフトウェアを紹介してくれています。
2021年8月現時点でこれらをまとめてみますと、DICOMからNIfTIに変換するソフトウェアとして以下の3つがメジャーなようです。どのソフトウェアもGUIで操作できるので簡単に使用できます。SPMはMATLABが必要なので有料ですが、それ以外はフリーのソフトウェアです。
このように多くの便利なソフトウェアがありますが、再現可能性を追求している私はPythonだけでファイル形式変換から解析までを一貫して行いたいと考えました。Pythonのパッケージがあれば実現できそうだと思い探してみたところ、dicom2niftiというパッケージを見つけました。
githubのコミット状況を見てみると現在も開発は継続しているようにみえます。今回の記事ではdicom2niftiを用いたファイルフォーマットの変換を試みたいと思います。
方法
動作環境と使用したデータ
インストール方法
anacondaでは以下のコマンドでインストールできます。
conda install -c conda-forge dicom2nifti
実行方法
Pythonスクリプトから実行
githubのREADMEに実行方法が紹介されています。以下の2行で変換できます。
import dicom2nifti dicom2nifti.convert_directory(dicom_directory, output_folder, compression=True, reorient=True)
dicom2nift.convert_directoryの引数は
- dicom_directory
- 変換するDICOMファイルが含まれるdirectory
- output_folder
- 出力先のdirectory
- compression (True or False)
- gzip圧縮をするかどうか
- reorient (True or False)
- 画像の向きをLASに変換するかどうか
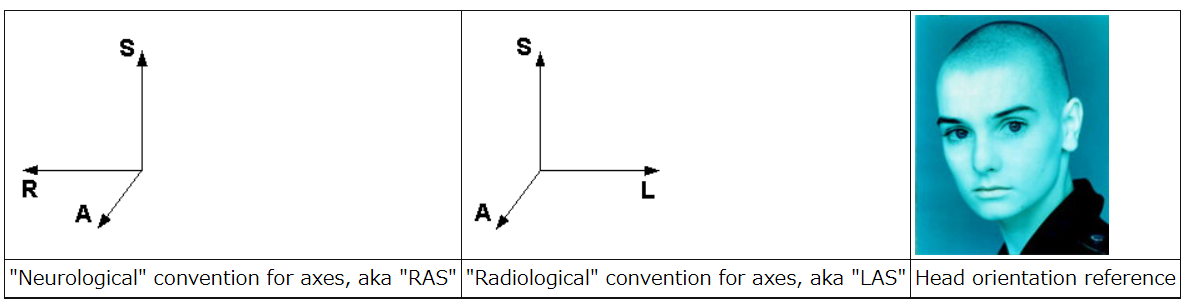
になります。僕ら「放射線科系」の人たちは画像の右側が対象の左側に対応している(LASと呼ぶらしい)のが通常の医用画像だと考えていますが、「神経科学系」の人たちは「放射線科系」の人が見ている画像の左右を逆にして表示すること(RASと呼ぶらしい)が通例なようです。画像の表示方法にも文化の違いがあるみたいですね。以下の画像がわかりやすいです。
コマンドラインから実行
コマンドラインからは以下のコマンドで実行できるようです。
dicom2nifti [-h] [-G] [-r] [-o RESAMPLE_ORDER] [-p RESAMPLE_PADDING] [-M] [-C] [-R] input_directory output_directory
各引数は
- -h (--help)
- ヘルプメッセージを表示
- -G (--allow-gantry-tilting)
- (CTの)ガントリーの傾けたデータの変換を可能にする
- -I (--allow-inconsistent-slice-increment)
- 一貫性のないスライスインクリメントデータの変換を可能にする
- -S (--allow-single-slice)
- シングルスライス(2D画像)の変換を可能にする
- -r (--resample)
- ガントリーが傾いたデータを直交画像に再サンプルしたり、スライス増分が一致しないデータを均一な画像に再サンプルする。
- -o RESAMPLE_ORDER (--resample-order RESAMPLE_ORDER)\
- リサンプリング時に使用するスプライン補間の次数 (0 -> 5) [0 = NN, 1 = LIN, ....]
- -p RESAMPLE_PADDING (--resample-padding RESAMPLE_PADDING)
- リサンプリング時に使用されるパディング値を塗りつぶし値として使用する
- -M (--allow-multiframe-implicit)
- implicit vr転送構文によるマルチフレームデータの変換を可能にする(動作保証はしていません)
- -C (--no-compression)
- gzip圧縮を無効にして、.nii.gzではなく.niiファイルを書き込む。
- -R (--no-reorientation)
- 画像のreorientationを無効にする(デフォルト:画像はLASの方向に再配置される)
のようです。こちらの詳細は詳しく書きませんが、githubを確認すれば書いていると思います。
windows環境でコマンドラインから実行する際は以下のissueが参考になりました。
簡単に説明すると、anaconda powershell promptを開いて仮想環境のdirectoryに移動して以下のコマンドを入力します。
mv .\Scripts\dicom2nifti .\Scripts\dicom2nifti_tool
その後、コマンドを実行する際は以下の通り入力します。
.\python.exe .\Scripts\dicom2nifti_tool --help # or other parameters`
結果
pythonスクリプトおよびコマンドラインからの実行はどちらもエラーがひとつでましたが、うまく変換できました。
エラーはdicom2nifti.exceptions.ConversionValidationError: TOO_FEW_SLICES/LOCALIZERと表示され、「シリーズ枚数が少ない画像はNIfTIに変換できませんでした」というエラーのようです。今回使用したデータのなかにシリーズが3枚のlocalizerがあり、それが引っ掛かったようです。ソースコードを見てみるとシリーズが3枚以下のシーケンスのデータはNIfTIに変換されない仕様になっているみたいです。少し探しましたが、これを回避する方法は見当たりませんでした。基本的に解析に使用する頭部の画像が4枚以下になることはないので、大きな問題はないと思います。
2021.8.29追記
dicom2nifti.disable_validate_slicecount()を実行しておくことで、3枚以下のシーケンスのデータもNIfTIに変換できるようになるようです。追記終わり。
2021.12.15追記
上のコードではダメでした。
import dicom2nifti.settings as settings settings.disable_validate_slicecount()
こちらのコードが正解です。追記終わり。
引数reorientをTrueにしたところ元の画像の上下が逆になりました。今回検証できたのはもともとがLASの画像に対してreorientをTrueにしても上下が逆になるだけで、Falseにすると元の向きのままだったという点のみです。元のDICOMがRASの場合であればLASに変わるのかもしれませんが、そのような画像が手元にないので検証できませんでした。
ひとこと
dicom2niftiはざっと調べたところDICOMからNIfTIへ変換できる唯一のPythonパッケージのようです。
(追記終わり)
pythonスクリプトは2行で変換できるので簡単でいいですね。
しかし、dcm2niixもしくはMRIconvertを使ったほうが自由度が高そうです。これらはNIfTIで出力する際のファイル名やdirectory構造を操作できるので、変換後のデータの整理の手間が省けそうです。なおMRIconvertはwindowsでは32bit版しかないので多くのファイルの処理をする際にはもしかしたらdcm2niixのほうが動作が早いかもしれません(これは想像です)。
まとめ
DICOMからNIfTIに変換するソフトにはdcm2niixもしくはMRIconvertを使うのがベターかもしれません。Pythonだけでどうにかしたいのであればdicom2niftiを使うのもいいかもしれません。